
OCHEM - Unsere Plattform zur Erstellung von in silico ADME/Tox Vorhersagemodellen
OCHEM ist eine Plattform für Medizinische Chemiker, Toxikologen und Chemieinformatiker.
Mit ihrer Hilfe lassen sich hochpräsize Modelle für Substanzeigenschaften - Quantitative Strutur-Eigenschafts- und Struktur-Aktivitätsmodelle (QSPR/QSAR) effizient generieren.
OCHEM ist eine einzigartige und umfassende Software-Suite, das Produkt jahrzehntelanger Arbeit und Erfahrung, die in einem einzelnen Produkt eine große Zahl an Software-Komponenten vereint.
Die Plattform ist eine Weiterentwicklung des Virtual Computational Chemistry Laboratory (VCCLAB).
Die VCCLAB Website hat derzeit mehr als 5.000 Besucher Monatlich, auf ihr werden jährlich mehr als 136.000 Analysen durchgeführt.
OCHEM integriert eine Vielzahl von maschinellen Lernverfahren, darunter alle Methoden, die wir verwendet haben, um die "ePhysProp"-Suite, zu entwickeln - d.h. die gleichen Algorithmen, mithilfer derer wir die Leistungsfähigkeit unserer Software unter Beweis gestellt haben. Details finden sich auf der ePhysProp Seite.
Fallbeispiele
OCHEM hat seine Qualität in einer Reihe von Studien bewiesen. Ohne Anspruch auf Vollständigkeit seien hier beispielhaft angeführt : Vorhersage der Komplexierungskonstanten (Tetko et al, 2006), Schmelzpunkte (Varnek et al, 2007), Toxizität (Zhu et al, 2008), mehrere Eigenschafsvoraussagen (Varnek et al, 2009), Mutagenität nach dem AMES-Test (Sushko et al, 2010), CYP450-Hemmung (Novotarskii et al, 2010) sowie einer Studie zur genaue Abschätzung der Anwendbarkeitsdomäne von Modellen (Tetko et al, 2009).Mehr über OCHEM
Experimentelle Daten
In OCHEM ist eine Datenbank von Molekülen zusammen deren ADMET Eigenschaften enthalten. Die einzigartige Struktur und erweiterten Funktionen unserer Datenbank erlauben die Speicherung von Daten mit den jeweilig zugehörigen Referenzen, so dass sie auf allen interaktiven Grafiken die Datenquelle mit einem Click verifizieren können. Da die Daten wie in der Referenz angegeben gespeichert werden, übernimmt OCHEM die automatische Konvertierung von Maßeinheiten. OCHEM hat vielerlei Schnittstellen, beispielsweise lassen sich Daten entweder aus Dateien (etwa per Excel- oder SDF-Format) oder über Web Services, z.B. im Verbund mit Workflowtools wie KNIME einspielen.
Die Bearbeitungsfunktionen erlauben das gleichzeitige Editieren von Molekülsätzen und die Plattform beinhaltet eine Änderungsverfolgung. Wir unterstützen unterschiedliche Benutzerrechte, die automatische Überprüfung von Molekülnamen und vieles mehr.
Modelldaten
OCHEM beinhaltet desweiteren ein Modellierungsystem. Die Modelldatenbank umfasst lineare und nichtlineare Methoden zur Modellentwicklung und Abschätzung ihrer Vorhersagegenauigkeit.
Ein Fokus der Entwicklung ist die automatische Abschätzung der Anwendbarkeitsdomäne von Modellen und die Daten-Integration, z. B. die Entwicklung von Modellen für mehrere Eigenschaften gleichzeitig.
Modelle für Adsorption, Blut-Hirnschrankendurchdringung, Hautpenetration und viele andere biologische Aktivitäten befinden sich derzeit in der Entwicklung.
Die Datenbank enthält zusätzlich zu den von unserem Team entwickelten Modelle eine Vielzahl von Modellen, die von Wissenschaftlern mit Hilfe unserer öffentlichen Version dieses Tools entwickelt wurden.
Durch die Integration all dieser Modellen stellt OCHEM ein Referenz-System für Modellvergleiche dar. Sowohl Web-Benutzer als auch Unternehmen haben die Möglichkeit, ihre Ansätze und Modelle einfach, schnell - und sicher vor Implementierungs- und Reproduktionsfehlern - mit veröffentlichten Methoden und Modellen zu vergleichen.
Die Nutzung von eigenschaftsbasierten Ähnlichkeitsfunktionen ist insbesondere für die Auswahl geeigneter Verbindung im frühen Hochdurchsatz (HTS)-Screening nützlich. Das Programm ermittelt automatisch Regionen im chemischen Raum, in denen die Vorhersagen ungenau sind und in denen die Modelle unsachgemäße Aussagen über die "druglikeness" von Verbindungen treffen. Dies ist besonders nützlich, um (virtuelle) chemischen Bibliotheken zu analysieren.
Wir bieten eine solche in-silico Analyse auch als Dienstleistung an.
Schnappschüsse


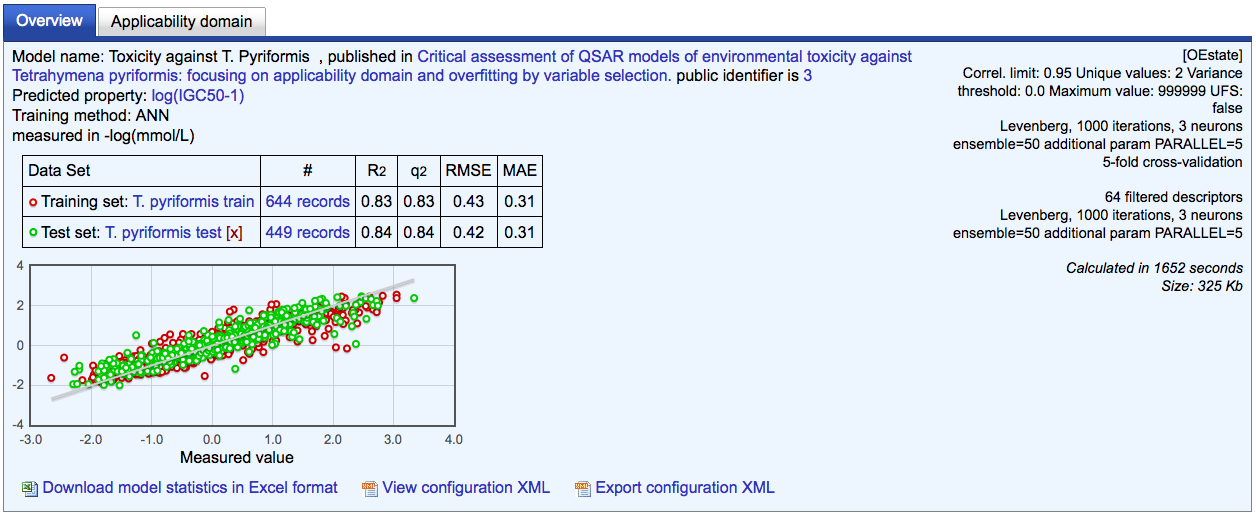
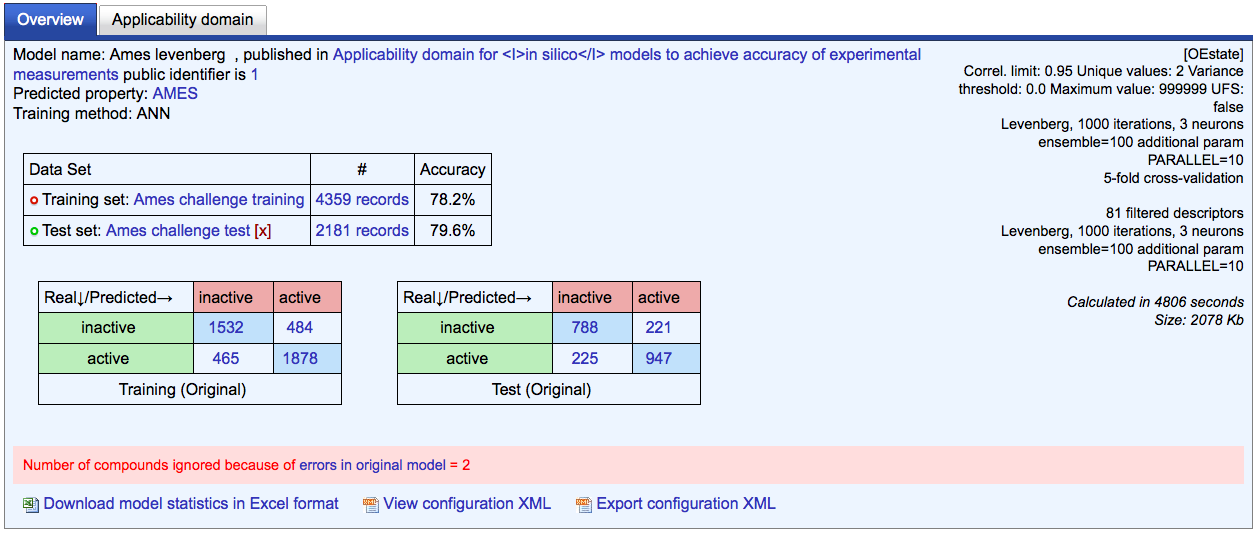
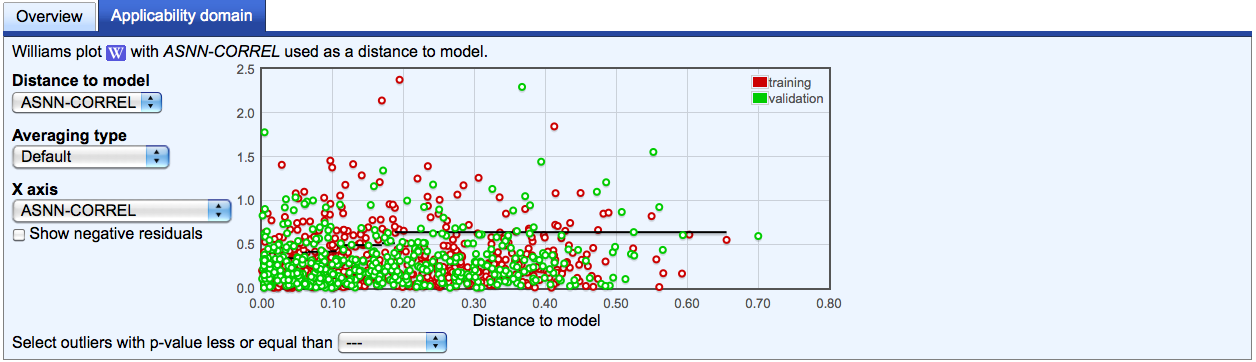
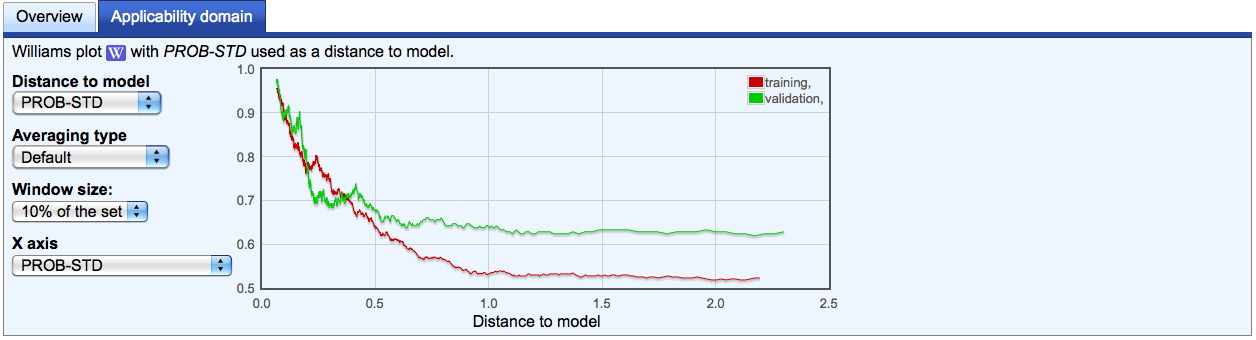
Lernen Sie OCHEM kennen
Wie passt OCHEM in meine bestehende Infrastruktur?
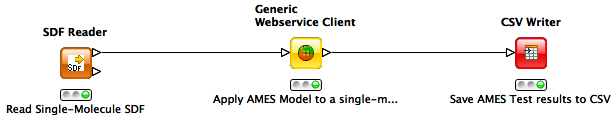
OCHEM bietet eine Vielzahl von Schnittstellen zu ihrer bestehenden Infrastuktur. Sie können OCHEM beispielsweise über die OCHEM Web Services in Ihre bestehende IT-Infrastruktur integrieren.|
Probieren Sie es aus - hier können Sie z.B. einen einfachen KNIME Workflow zum Abschätzen der AMES-Aktivität eines einzelnen Moleküls herunterladen. Wir stellen Ihnen gerne ein entsprechendes Beispiel für Ihre Anwendung zur Verfügung oder integrieren OCHEM für Sie in Ihre Infrastruktur. |
|
Präsentationen
- OCHEM Präsentation beim Chemaxon User Group Meeting 2011 (Englisch)
- Use Case : Wie kann ich mit Hilfe von OCHEM Informationen über das Ergebnis eines AMES-Tests bekommen? Beispiel : Captopril (Deutsch)
Versionen
OCHEM Academia
OCHEM Academia ist eine öffentliche, frei zugängliche Onlineversion für akademische Nutzer.
OCHEM Lite
OCHEM Lite ist eine Einzelplatzversion. Unsere Standardinstallation basiert auf einem 27'' iMac, der unter Ubuntu betrieben wird. Wir liefern Ihnen einen komplett installierten Server incl. einem Tag Service für Einführung und Integration in Ihre IT-Umgebung.
OCHEM Flex
Die konfigurierbare OCHEM Standardversion. Wir richten OCHEM auf einem Ihrer Server ein oder liefern Ihnen einen komplett installierten Server. Haben Sie einen eigenen Rechencluster oder ein bewährtes 3D-Optimierungsverfahren ? Wir integrieren OCHEM für Sie in die bestehende Rechnerinfrastruktur. Die maximale Größe des Rechenclusters bestimmen Sie - und sie lässt sich bei Bedarf derzeit in Minuten anpassen. Wir liefern incl. zwei Tagen Service für Einführung und Integration in Ihre IT-Umgebung.
OCHEM Enterprise
Die OCHEM Enterprise Edition ist unsere Version für Großunternehmen. Ihr Einsatz unterliegt keinerlei Beschränkungen. Die Version beinhaltet einen kompletten Technikermonat Installationssupport plus alle andere notwendige Arbeiten, um eine unternehmensweite Einführung und die Eingliederung in die bestehende Infrastruktur sicherzustellen.
In der Entwicklung: Das "ÖkoTox Assess & Report"-Paket
Das "ÖkoTox Assess & Report"-Paket wird OCHEM für die Beurteilung der ökotoxikologischen Auswirkungen von kleinen Chemikalien ergänzen. Im Rahmen dieses Paketes entwickeln wir Modelle zur Abschätzung REACH-relevanten Endpunkten. REACH ist das neue europäische Chemikalienrecht. Die REACH-Verordnung wurde am 30. Dezember 2006 im Amtsblatt der Europäischen Union Nr. L 396 als Verordnung (EG) Nr. 1907/2006 veröffentlicht und löst zahlreiche bestehende gesetzliche Regelungen ab.
Im Rahmen des "ÖkoTox Assess & Report" entstehen u.A. Modelle für- Physikalisch-chemische Eigenschaften: Schmelz-und Siedepunkt, Dampfdruck, Kow, Koc und Wasserlöslichkeit
- Umweltverträglichkeit: Biologischer Abbau, Hydrolyse, Photolyse, Atmospherische Oxidation und Bioakkumulation
- Ökologische Wirkungen: Aquatische Toxizität: Akut-und langzeittoxizität für Fische, Daphnien und Algen
- Akut-und Langzeit- Sedimenttoxizität
- Akute und langfristige terrestrische Effekte
- Das Marie Curie initial training network “Environmental Chemoinformatics” (ECO), welches durch eADMETs wissenschaftlichen Leiter, Dr. I.V. Tetko, koordiniert wird
- Case studies on the Development and Application of in-Silico Techniques for Environmental hazard and Risk assessment (CADASTER)
In der Entwicklung: Das "iPRIOR" Paket für die Toxizitätsvorhersage
iPRIOR dient der Abschätzung der in vivo Toxizität von Verbindungen durch die Analyse ihrer Wechselwirkungen mit toxikologischen Pathways in in-vitro-Experimenten in Verbindung mit Experimentellen und Vorhergesagten physikalisch-chemischen (z. B. Löslichkeit, Lipophilie, Dissoziation Zustand) und biologischen (z. B. Bioverfügbarkeit, metabolische Stabilität) Eigenschaften.
Diese Forschungsaktivitäten werden von uns im Rahmen des BMBF-Forschungsprogramms "GO-Bio" durchgeführt.
